AI-metoder hittar nya celltyper i vävnad
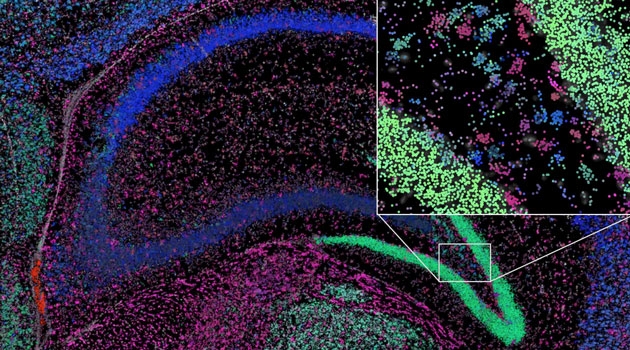
Med så kallad in situ-sekvensering är det möjligt att skapa mikroskopibilder av genaktivitet direkt i vävnad. För att lättare kunna tolka den stora mängd information som då fås har forskare vid Uppsala universitet nu utvecklat en helt ny bildanalysmetod, baserad sig på AI-algoritmer för nätverksanalys som ursprungligen togs fram för att förstå sociala nätverk. Studien är publicerad i tidskriften FEBS Journal.
Vävnaden som bygger upp våra organ består av miljontals celler med olika funktion. Alla celler hos en individ har samma gener (DNA) som finns i cellkärnan. Generna uttrycks genom att molekyler kallade mRNA, eller budbärar-RNA skickas ut från cellkärnan till övriga cellen med information om vad som ska göras. Kombinationen av mRNA definierar på så sätt cellens funktion och identitet.
informationsteknologi vid Uppsala universitet.
Foto: David Naylor
RNA kan avläsas med hjälp av in situ-sekvensering, en metod som forskare bakom den nya studien tidigare varit med och utvecklat och där miljontals avlästa mRNA syns som prickar i en mikroskopibild av vävnaden. Problemet är att de kan vara svårt att urskilja alla viktiga detaljer. Det är där den nya AI-baserade metoden kan vara till nytta. Med den är det nämligen möjligt att läsa av både funktioner inom en enskild cell och interaktioner mellan celler.
– Vi utnyttjar de senaste metoderna inom AI, eller mer specifikt grafbaserade neurala nätverk som utvecklats för att analysera sociala nätverk och anpassar den för att förstå biologiska mönster och gradvisa variationer i vävnadsprover. Man kan likna cellerna vid sociala enheter som liksom grupper av individer kan definieras utifrån de aktiviteter de delar inom sina sociala nätverk som till exempel twitter, sökningar på google eller val av tv-serier, säger Carolina Wählby, professor vid institutionen för informationsteknologi vid Uppsala universitet.
Kräver ingen förkunskap om förväntade celltyper
Tidigare analysmetoder av den här typen av data är beroende av att det redan är känt vilka celltyper som finns i vävnaden och att cellkärnorna i den är identifierade. Metoden som brukar användas kallas singel-cell-analys, men den kan tappa bort en del mRNA och missa vissa celltyper. Trots avancerad automatiserad bildanalys är det ofta svårt att hitta de olika cellkärnorna om till exempel cellerna ligger tätt.
– Med vår analys som vi kallar spage2vec kan vi nu få fram motsvarande resultat utan någon förkunskap om förväntade celltyper och kan dessutom hitta nya celltyper och funktioner inom eller mellan celler i vävnaden, säger Carolina Wählby.
Undersöker hjärtats tidiga utveckling
Forskargruppen arbetar nu vidare med sin analysmetod genom att undersöka hur olika celltyper uppstår och organiserar sig under hjärtats tidiga utveckling. Det är ren grundforskning som syftar till att öka kunskapen om de mekanismer som styr utvecklingen, både när allt fungerar som det ska och vid sjukdom.
I ett annat projekt som görs tillsammans med cancerforskare hoppas de kunna använda metoderna för att bättre förstå hur tumörvävnad på molekylärnivå interagerar med intilliggande frisk vävnad. Målet är att det på sikt ska kunna leda till bättre behandlingar som anpassas efter varje patient.
Åsa Malmberg
Publikation
Referens: G. Partel, C. Wählby (2020); Spage2vec: Unsupervised representation of localized spatial gene expression signatures, The FEBS Journal. DOI: 10.1111/febs.15572